
Photo by Matthew Tan on Unsplash
RのHistDataパッケージのCholeraのデータは、1948年から49年にかけての英国のコレラによる死亡者のデータです。
コレラの原因が何なのかを調べるデータです。
まずは、データを呼び出してstr()関数を使ってみます。

38の観測と15の変数を持つデータフレームです。
ヘルプのコードはplot()関数でcholera_drateとelevationの散布図を描いています。
cholera_drateは1848年の人口10,000人当たりのコレラの死亡者数です。
elevationはhigh warter markからの高さ(フィート)です。


elevationが小さいほうが死亡率が高いように見えます。
次のコードは、cholera_drateがelevationに反比例している曲線を追加しています。


次はcarライブラリーのscatterplot関数でもう少し見栄えのいい散布図を描いています。

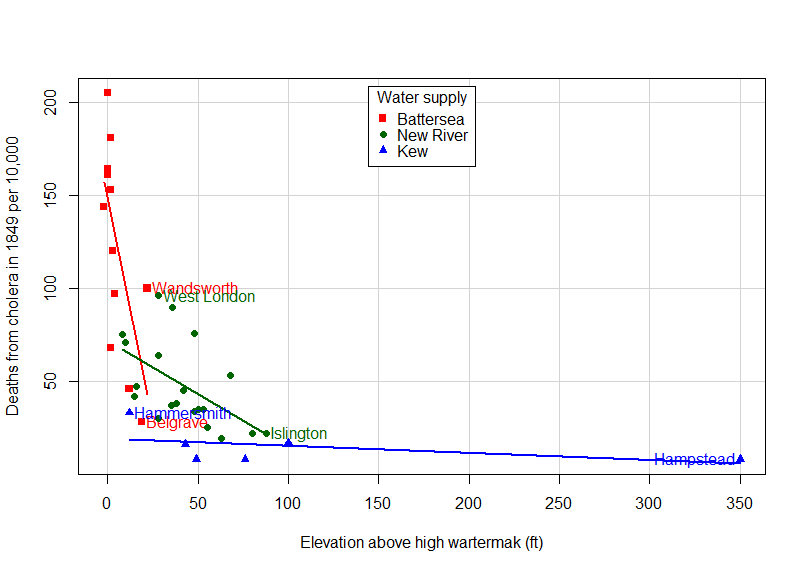
water: 水の供給地区 別に色分けして散布図を描いでいます。
もう一つの散布図のコードは、


poor_rate: 貧困率とコレラ死亡者率の散布図です。貧困率が高いほど死亡率が高いです。
次はglm()関数でロジスティクス回帰分析をしています。

最後のコードはOR、オッズレシオを計算しています。

そして最後にeffectsパッケージのallEffects関数を使っています。

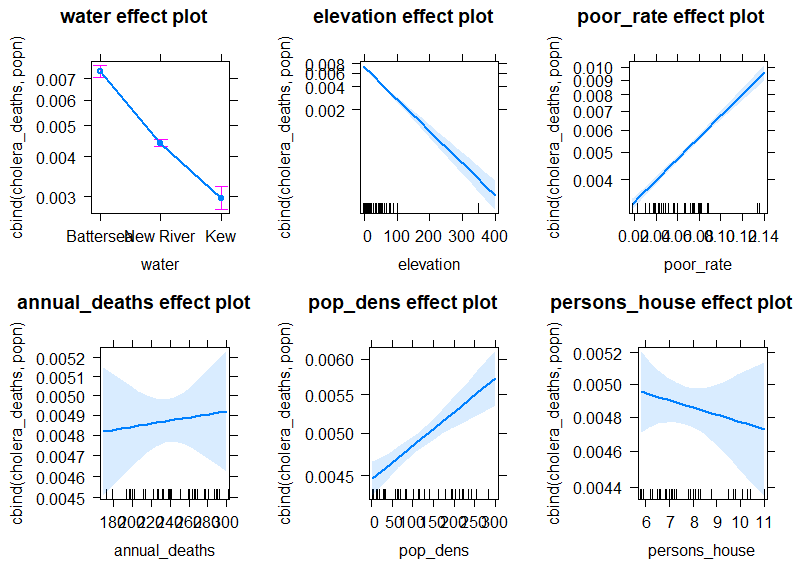
これでおしまいです。
以下は今回のコードです。
# 2022-02-11
# HistData - Cholera
#
library(HistData)
data("Cholera")
#
str(Cholera)
#
# plot cholera death vs. elevation
plot(cholera_drate ~ elevation, data = Cholera,
pch = 16, cex.lab = 1.2, cex = 1.2,
xlab = "Elevation above high wartermark (ft)",
ylab = "Deaths from cholera in 1849 per 10,000")
#
# Farr's mortality ~ 1 / elevation law
elev <- c(0, 10, 30, 50, 70, 90, 100, 350)
mort <- c(174, 99, 53, 34, 27, 22, 20, 6)
lines(mort ~ elev, lwd = 2, col = "blue")
#
# better plots, using car::scatterplot - wartermark vs. drate
if (require("car", quietly = TRUE)) {
# show separate regression lines for each water supply
scatterplot(cholera_drate ~ elevation | water, data = Cholera,
smooth = FALSE, pch = 15:17,
id = list(n = 2, labels = sub(",.*", "", Cholera$district)),
col = c("red", "darkgreen", "blue"),
legend = list(coords = "top", title = "Water supply"),
xlab = "Elevation above high wartermak (ft)",
ylab = "Deaths from cholera in 1849 per 10,000")
}
#
# better plots, using car::scatterplot - poor rate vs. drate
if (require("car", quietly = TRUE)) {
scatterplot(cholera_drate ~ poor_rate | water, data = Cholera,
smooth = FALSE, pch = 15:17,
id = list(n = 2, labels = sub(",.*", "", Cholera$district)),
col = c("red", "darkgreen", "blue"),
legend = list(coords = "topleft", title = "Water supply"),
xlab = "Poor rate per pound of house value",
ylab = "Deaths from cholera in 1849 per 10,000")
}
#
# fit a logstic regression model a la Bingham etal.
fit <- glm(cbind(cholera_deaths, popn) ~ water + elevation + poor_rate +
annual_deaths + pop_dens + persons_house,
data = Cholera, family = binomial)
summary(fit)
#
# odds ratios
cbind(OR = exp(coef(fit))[-1], exp(confint(fit))[-1, ])
#
if (require(effects)) {
eff <- allEffects(fit)
plot(eff)
}
#