
Bing Image Creator で生成: Close up of Helleborus argutifolius flowers, background is snow mountains and green woods, photo
の続きです。今回はランダムフォレストのモデルを作ります。
まず、コアの数を確認します。

私のラップトップは、コアの数は8個でした。
次は、ランダムフォレストのモデルを設定します。

ランダムフォレストには、mtry, trees, min_n という3つのパラメータがあります。このうち、trees だけ1000に決めて、他はチューニングでベストな値を決めます。
続いて、レシピの設定をします。
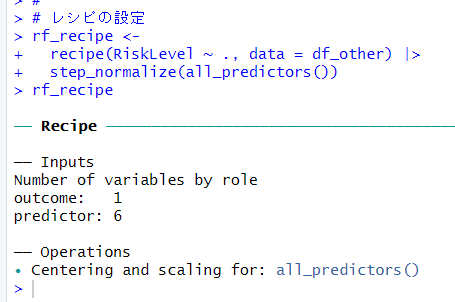
前回の LASSO回帰と同じく、変数は標準化しておきます。ランダムフォレストは本当はその必要な無いようですが、とりあえずやっておきます。
そして、ワークフローを設定します。

パラメータチューニングをします。

show_best() で最適なパラメータを確認します。

mtry = 6, min_n = 5 が mean が 0.986 と一番大きな値です。
autoplot() でグラフを描きます。


select_best() でベストモデルを特定します。

ROC カーブを描きます。


LASSO 回帰の ROC カーブよりもよさそうですね。
LASSO 回帰の ROC カーブと重ねて表示します。

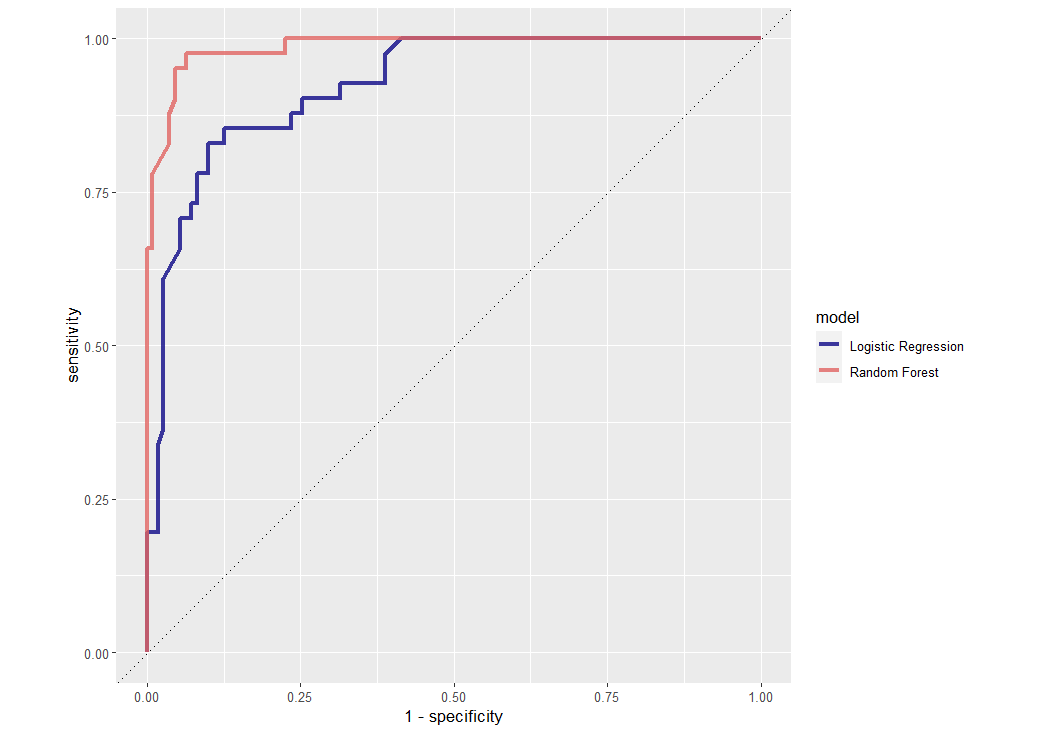
ランダムフォレストのほうが優れています。
mtry = 6, min_n = 5 で最終モデルを設定します。

最終ワークフローを設定します。

レシピは同じなので、モデルだけを更新しています。
モデルをフィットさせます。

このモデルの評価指標をみてみます。

accuracy が 0.925 なので、かなり正確に予想できるようです。
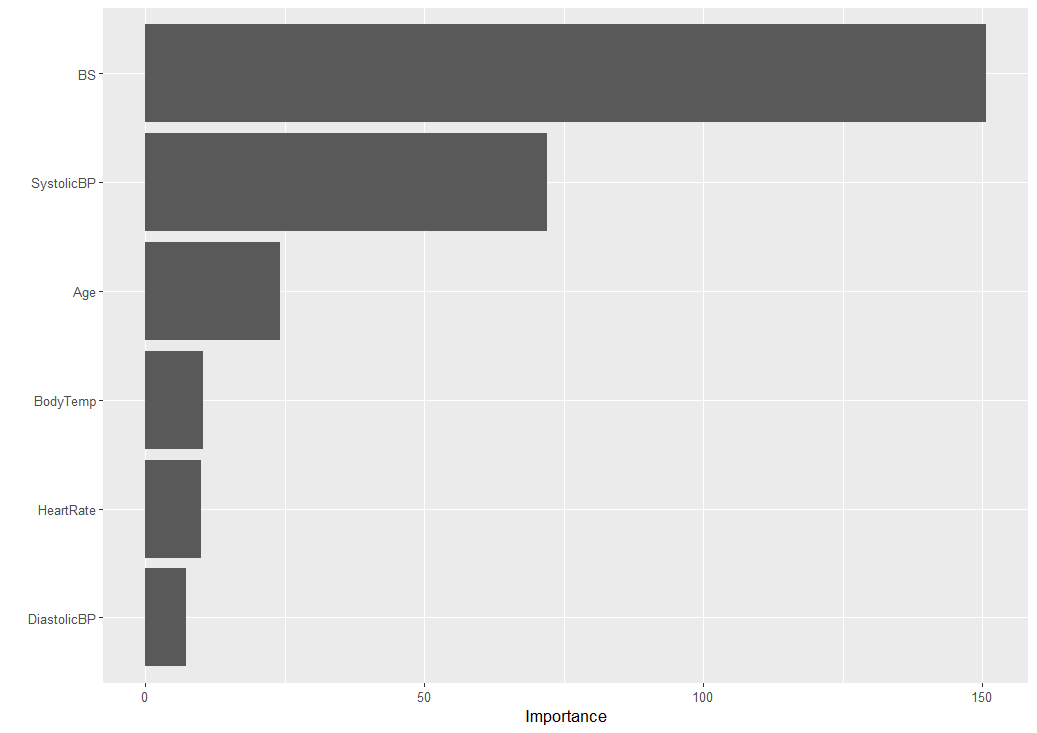
どの変数が重要なのかみてみます。

ROC Curve を描いてみましょう。


df_other のデータでモデルをフィットさせて、df_test のデータの予測をします。

予測結果をみてみます。
正解率は0.9249です。LASSO 回帰は、0.806 でしたのでかなり優れています。
high risk だけの正解率は、

0.838 とこちらも LASSO 回帰の 0.382 と比べると大幅に向上しています。
さすが、ランダムフォレストですね。
今回は以上です。
初めから読むには、
です。
今回のコードは以下になります。
#
# コアの数を確認
cores <- parallel::detectCores()
cores
#
# Random Forest Model
rf_mod <-
rand_forest(mtry = tune(),
min_n = tune(),
trees = 1000) |>
set_engine("ranger", num.threads = cores) |>
set_mode("classification")
rf_mod
#
# レシピの設定
rf_recipe <-
recipe(RiskLevel ~ ., data = df_other) |>
step_normalize(all_predictors())
rf_recipe
#
# ワークフローの設定
rf_workflow <-
workflow() |>
add_model(rf_mod) |>
add_recipe(rf_recipe)
rf_workflow
#
# パラメータチューニング
set.seed(111)
rf_res <-
rf_workflow |>
tune_grid(val_set,
grid = 25,
control = control_grid(save_pred = TRUE),
metrics = metric_set(roc_auc))
#
# トップモデル
rf_res |>
show_best(metric = "roc_auc")
#
# グラフ
autoplot(rf_res)
#
# ベストモデルを特定
rf_best <-
rf_res |>
select_best(metric = "roc_auc")
rf_best
#
# ROC Curve
rf_auc <-
rf_res |>
collect_predictions(parameters = rf_best) |>
roc_curve(RiskLevel, .pred_high_risk) |>
mutate(model = "Random Forest")
autoplot(rf_auc)
#
# Logistic Regression と Random Forest の ROC Curve
bind_rows(rf_auc, lr_auc) |>
ggplot(aes(x = 1 - specificity, y = sensitivity, col = model)) +
geom_path(lwd = 1.5, alpha = 0.8) +
geom_abline(lty = 3) +
coord_equal() +
scale_color_viridis_d(option = "plasma", end = 0.6)
##
# 最終モデル
last_rf_mod <-
rand_forest(mtry = 6,
min_n = 5,
trees = 1000) |>
set_engine("ranger",
num.threads = cores,
importance = "impurity") |>
set_mode("classification")
#
# 最終ワークフロー
last_rf_workflow <-
rf_workflow |>
update_model(last_rf_mod)
#
# 最終フィット
set.seed(111)
last_rf_fit <-
last_rf_workflow |>
last_fit(splits)
last_rf_fit
#
# 評価指標
last_rf_fit |>
collect_metrics()
#
# Variable Importance
last_rf_fit |>
extract_fit_parsnip() |>
vip()
#
# ROC Curve
last_rf_fit |>
collect_predictions() |>
roc_curve(RiskLevel, .pred_high_risk) |>
autoplot()
#
# Final Model
set.seed(111)
final_model <- last_rf_workflow |>
fit(df_other)
#
# df_test のデータセットで予測
predictions <- predict(final_model, df_test)
predictions
#
# 予測結果: Contingency Table
table(df_test$RiskLevel, predictions$.pred_class)
#
# 正解率
(57 + 177) / (57 + 177 + 11 + 8)
#
# high_risk の正解率
57 / (57 + 11)
#